[ 27: 腸管出血性大腸菌の病原性制御因子による遺伝子発現制御機構 ]
O-157 のゲノムはモザイク構造。
いろいろな外来性遺伝子がはいっているために場所によって GC 含量が違う、とか。
では、外来性遺伝子は病原性を発揮するためにどのように制御されているのか?
ふつうは大腸菌の H-NS によって外来性遺伝子が silencing されており、これを knockout すると O-157 でも病原性があがる。
Pch がある・ない条件で、H-NS がどこにくっついているかを調べた。
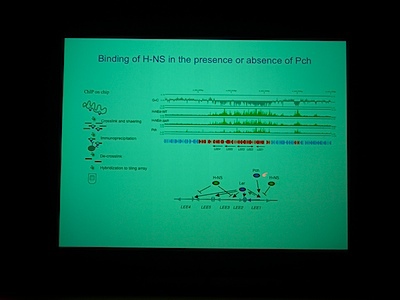

H-NS と Pch の binding site は非常によく似ている。Pch+ でも H-NS の bind する量はあまりかわらないが、よくみると変わっている (H-NS の bind が若干下がる) ところがあり、そこにはLEE1-5 などの遺伝子がコードされている。
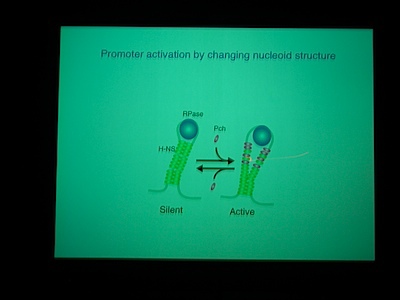
PchA の発現量を増やしてやると、LEE の発現量も増える→病原性上がる。
Pch がついているときは、H-NS が小さな固まりで bind している模様。H-NS がいっぱいくっついているときは転写を阻害しているが、Pch がはいってきちゃうと転写されるようになっているのではないか。
Pch は病原性遺伝子だけを制御している? → ほかのところにもつきます。あと、H-NS や Pch 以外のファクタもあると思う。
H-NS や Pch がつく場所のコンセンサス配列みたいなのはわかるの? → AT rich だということくらいしかいえない。タンパクのサイズは 10kd くらい。
[ 28: 枯草菌の複数シグマ因子による転写制御ネットワーク解明の試み ]
たくさんのシグマ因子がたくさんの遺伝子の調節をこまかくやっている。
ECF family が半分くらいあるといわれているが、機能が (環境応答らしい、ということくらいしか) よくわからない場合が多い。発現ストレスが似ていたり、プロモータ領域が似ていたりして、つぶして解析したりすることがむずかしい。
7つあるので、それをまず全部破壊する (抗生物質耐性の遺伝子とかがあるので、マーカーありでやるのは難しい)。
WT でも、胞子形成後に溶菌する。
ECF7 つと、sigI を壊すと溶菌する率があがることがわかった。
いくつかの ECF と sigI を破壊して、同じような様相を呈するケースを探している。
– 溶菌は2成分制御系 (YvrG/ YvrHb とかで、最終的にオートリシン LytC が発現)。
– で、yvrG は sigI に制御されているので、sigI を壊すと最終的に溶菌の制御も壊れる。
– yvr* を IPTG で転写誘導したり、lytC を壊すことで溶菌は抑圧できる。
ECF family は環境応答だけでなく恒常性維持とかにも関わっているらしい。
大腸菌の programmed cell death との関わりは? → やっぱりシグマ因子だけど、大腸菌ではシグマ因子の発現が細胞死を誘導するので、逆になっている。
[ 29: 枯草菌におけるフラボノイド応答性転写制御系の機能解析 ]
気絶。すみません…
[ 30: シアノバクテリアSynechococcus elongatus PCC 7942 における二成分制御系シグナル伝達ネットワークの解析 ]

多くの場合は2成分制御系のヒスチジンキナーゼとレスポンスレギュレータはゲノム上に並んで operon 構造を持っているが、シアノバクテリアでは並んでおらず、orfan になっている。

Operonになっているものもあるが、orphan なものですべてのシアノバクテリアに保存されているものが 7 つ。NblS, RpaB, Syc1065, Ycf29 はシアノバクテリアの必須遺伝子であり、紅藻からも同定されている重要な遺伝子。
相互作用を網羅的に調べてみた。16遺伝子間、10組のパートナーを同定。ヒスチジンキナーゼ同士の組もある。
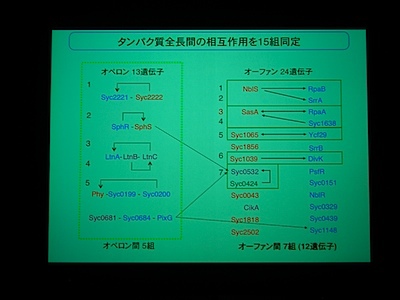
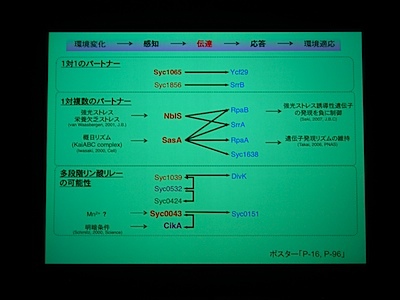
1対複数のパートナーとかを構成するためには operon より orfan でいたほうが都合がよいのかも? ということでした。
[ 31: Rhodobacter sphaeroidesの光合成から好気呼吸へのエネルギー代謝転換時における全ゲノム転写プロファイルの動的変化 ]
染色体2つとプラスミド5個。
嫌気性条件下で特異的に光合成 (自己栄養)。酸素があれば呼吸する。酸素のある・なしで定常条件になっている奴らは充分に研究されているが、transition state は?
Gene chip を使って、嫌気性条件から好気性条件へ遷移する transition state における発現をしらべた。NADH dehydrogenase なども嫌気性条件と好気性条件で別々の遺伝子を使ったりしているところがおもしろい。
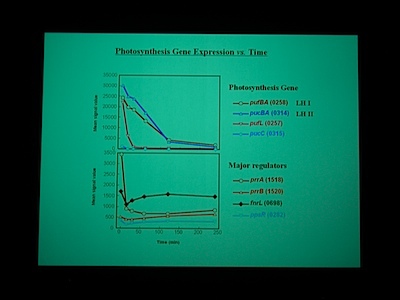
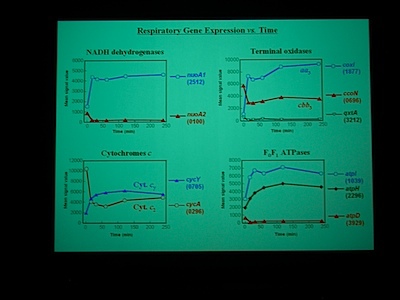
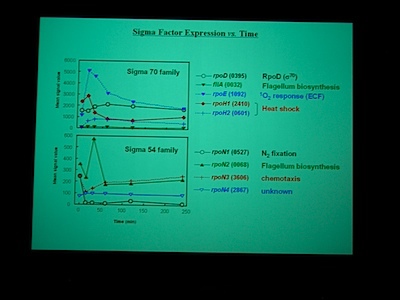
Class I – IV の遺伝子に発現をみていると、酸素を与えたところで細胞周期が同期している。
好気性条件になると、最初に光合成で作った糖を食べて、それから培地の炭素源を使う。
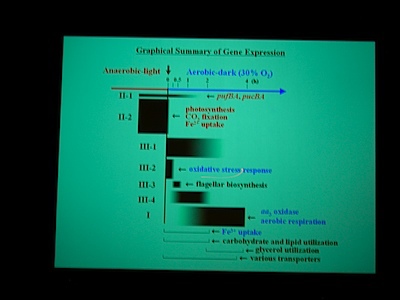
いろいろな生き物がいるねぇ・・・・
[ 32: 構造-機能解析によるシアノバクテリオクロムの多様性と普遍性の理解 ]
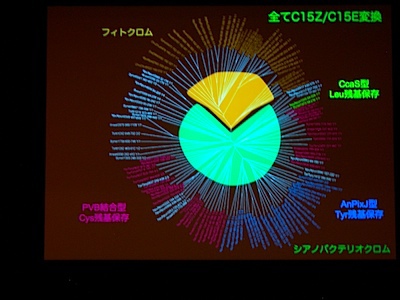
光受容体 (Phytochrom) に似た、シアノバクテリアにしかないシアノバクテリオクロムというのが注目されている。ものすごくたくさん種類があり、吸収スペクトルが違う。
AnPixJ の Pr 型を結晶化することに成功。フィトクロムの Pr 型と比べると、タンパク質と色素の位置関係が違うものの、タンパク質どうし、色素どうしはよく似ている。でも、色素のまわりの水素結合のようすはだいぶ違う。
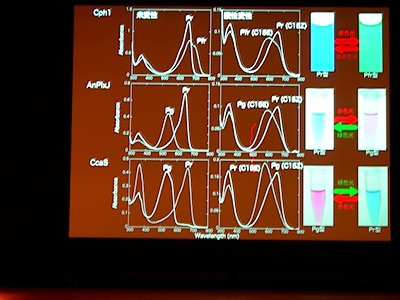
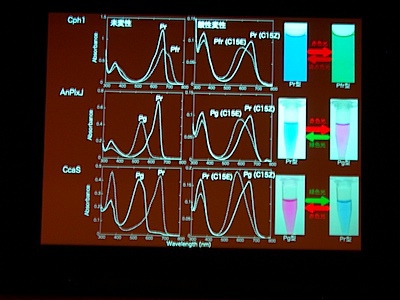
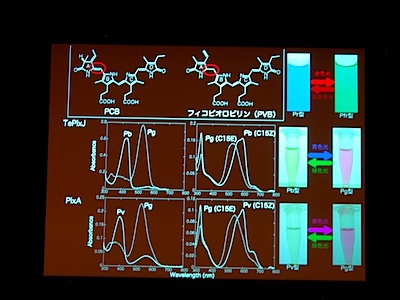
タンパク質と色素の共進化ですが、祖先型みたいなのはわかるかしら? → おもしろい suggestion ですが、とてもむずかしい。
ここまで多様な理由は? → シアノバクテリアは緑色光を吸収する必要があったりとか、そういうことが理由の一つとして考えられる。
月: 2008年3月
ゲノム微生物学会 Mar.07 / oral session 4
[ 21: Replication dynamics of the Vibrio chromosomes affect gene dosage, expression and location ]
ふたつある染色体のそれぞれの複製のときにどんな遺伝子がたくさん発現してるか、とかを実験的に確認。
片方の染色体は重要で安定しており、もう片方は flexible.
小さい方の origin は plasmid かも、というお話。

[ 22: プラスミド分配の目的地を規定するモータータンパク質、ParA ]
細胞分裂のときにプラスミドはどうやって分配されるのか?
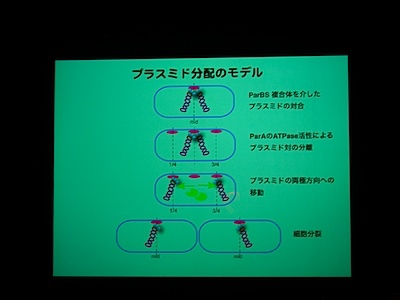
プラスミド分配に必要なタンパク質には actin type のものと walker type のものがある。
actin type はうにょーっと伸びるわけだが、walker type は細胞の極から極へ移動し、プラスミドを連れていくっぽい。
ParA protein に YFP, Plasmid に LacI promoter + CFP をつけて動態を観察。
ParA を追いかけると、細胞中央に輝点が現われ、1/4 地点に移動した後に 3/4 地点にも現れる。
プラスミドがふたつになる前に ParA がふたつになっている?



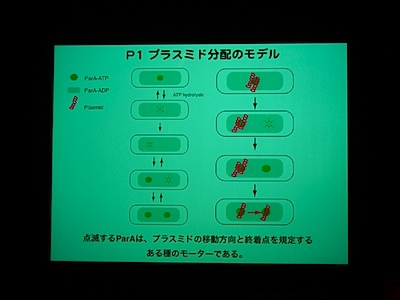
ParA を壊したり、ParA の ATPase を壊したりすると、局在しなくなる。
ParA を overexpression させたら? → 安定しなくなっちゃいます
[ 23: 枯草菌の50Sリボソームサブユニットの生合成におけるGTP結合タンパク質の結合モデルの提案 ]
50S subunit の後期生合成の関する研究。なんかすごい。
でも、完璧にウェットなお話でした。わからんです。
[ 24: Phylum Bacteroidetesに位置する細菌の菌体外タンパク分泌機構と滑走運動機構との関連性 ]
自分で糖発酵をせず、エネルギーを外界に依存。
表面に強力なプロテアーゼ (ジンジパイン: これ自体病原因子?) を持つ。たいていは膜結合な複合体になる。
分泌機構はよくわかっていないが、変異株にはジンジパインを外側に出すことができず、前駆体を細胞内に蓄積してしまうものがある。
P. gingivalis 輸送系に影響を与える変異を F. johnsoniae にいれてやると、滑走しなくなる。
膜タンパクであるところが共通だけど、それ以上のことについてはまだよくわからない。
[ 25: 分裂酵母Meu14は前胞子膜開口部を量依存的に制御する ]
分裂酵母の胞子形成。減数分裂後に核を包み込むように膜が生成される。
膜が袋状になるしかけが不思議。膜の先端には Meu14 というタンパク質があり、それが巾着の口をとじるところに関係している?
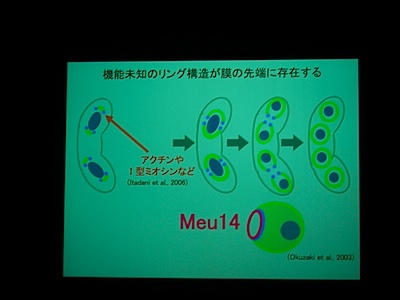
膜と核と Meu14 を蛍光で観察。
Meu14 がないと、開口が維持されず、さっさと口を閉じてしまって、核を包み込むことができなくなる。
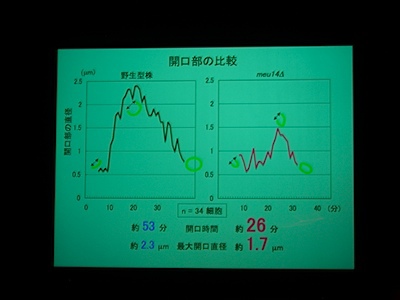
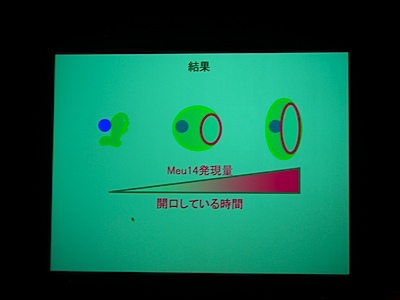
Meu14 を overexpression させると、口が大きく開き、開口している時間も長くなる (開くのにかかる時間は同じで、閉じるのに時間がかかる) が、まあ、わりとうまく動く。
開口は Meu14 の重合で、閉じるのは脱重合とすると、重合は濃度に依存し、脱重合は依存しないので、この現象が説明できる。
また、Meu14 は開口部をもたない膜には局在しないことが確認された。
脱重合のトリガになるような因子はなにかある? → まだわかっていません。
膜の開口部がどういう構造になっているかわかる?一重膜の開口部って? → あ、二重膜です。
動画を見ていると開口部どうしがおたがいにくっついて同調しているようにみえるんだけど → 相互作用しているかもしれないんだけど、近接している時間は非常に短いので、そうじゃないかも。離れていても同調して閉じます。
中身の核やなにかとの相互作用はないんですか?中身がちゃんとしたものであることを確認してから閉じているとかはない? → Meu14 がないと、核を分配するスピンドルが折れちゃったりするので、何かあると思う。
ゲノム微生物学会 Mar.07 / oral session 3
Bioinformatics のセッション。
[ 14: メディカルゲノム黄色ブドウ球菌タンデムパラログ遺伝子群における多様性形成モデル ]
バスに乗り遅れて、間に合わなかった…
[ 15: ゲノムアライメントに基づく近縁微生物ゲノムのコア構造の抽出 ]
基生研の内山先生。
Core = 類縁ゲノム感で主に垂直的に伝搬し、広く保存されている。
共通部分をとることで見つけるのが簡単。でも、
– 全部が持っている遺伝子は、生物種が増えるほど減る
– 正しいオーソログをどうとるか?
そこで、遺伝子の並び順の保存性に基づいて決定することにする。
MBGD で ortholog group を作成、グラフを作って DP で決定。

半分以上で保存されていればいいことにして、バチルス: 1376, 腸内細菌: 1814 のコア遺伝子セットを抽出。
ゲノムが大きいからといって、コア遺伝子を全部持っているとは限らず、コア構造からはたくさん欠失してしまっている場合もある。
並びはわりときれいに保存されている。

コア遺伝子には metabolism とか replication が多い。逆に膜輸送・signal transduction とかが少ない傾向。
RECOG というツールを公開予定。MBGD でできることがだいたいできる?
[ 16: 248種の原核生物ゲノム間の膜タンパク質割合 ]
SOSUI: 膜タンパク質予測ツール。
このツールを使った結果、総遺伝子数に占める膜タンパク質の割合はどんな生物種でもだいたい同じ、と予測。
多少ばらつきがあるが、ばらつきは正規分布でフィッティングできる。
ゲノムにランダムな変異を与えて (ただし、アミノ酸組成の割合は保つという制約あり) シミュレーションしてみると、同じ傾向のばらつきになる。
本質とは関係ないが、めちゃめちゃスライドがかっこいい。図とかが気合い入ってる。
Mac かと思ったら、Office XP なんだけどね。やっぱりツールよりセンスが重要ってことです。
[ 17: オリゴペプタイド情報を用いた一括学習型の自己組織化マップ法による機能未知のタンパク質類の機能推定法の開発 ]
SOM で、2連・3連アミノ酸の使用頻度を学習させることでタンパク質の機能を分類。
タンパク質はひとつひと機能とは限らない、とこいうところが今後の問題。
[ 18: 代謝ネットワークにおけるノードの「機能」とネットワーク内での「位置」の関係に関する研究 ]
完全に気絶してました。
[ 19: KazusaAnnotation: ゲノム情報への注釈付け、注釈の利用を支援するシステム ]
Social bookmark による open annotation.
http://a.kazusa.or.jp/
アノテーションと文献と関連づけとか、そういう感じ。どの文献にどの遺伝子がでてきたか、みたいなのを集積できるのだが、どうやら手動っぽい。
ふうむ。
でも、social bookmark 的にやれるのはおもしろいね。
[ 20: GenomeMatcher比較ゲノム用ソフトウエア ]
http://www.ige.tohoku.ac.jp/joho/gm.html
– BLAST, clustalW などを呼べる
– ユーザの注釈情報を簡単に反映できる
Excel からannotation が copy&paste で流し込める (フォーマットは決まってるけどね)。かっこいい!
拡大したりパラメータをかえて描いた絵を一覧にしておけるところがイカす。
gap のところを clustalW に投げたりできるのかー。
うおお。細かいところを見られるのがいいね。
丸いのも見られる。
やべー、俺もやることはまだまだあるな!
どれくらい大きい配列までいける? → 300Mbp くらいまで行ける。10Mbp くらいに区切って、1時間とか。
ゲノム微生物学会 Mar.06 / oral session 2
メタゲノムのセッション。
[ 8: 原油 n-アルカンの多様性と原油由来 DNA から推定される微生物集団のプロファイリング ]
原油から DNA を抽出して、もともとの生物を調べたい。
地球上の炭素のうち、生物がサイクルしているのはほんの一部で、その一部が石油になったりする。
原油のなかには原油を分解する菌とかも入っちゃうので、その中から大昔のゲノムを抽出するのは難しい。
原油は産出場所によって、アスファルト含有量とか粘土といった性質が違う。n-アルカンの diversity はその一つ。
中東産 (3種類例がでてた) のものはほぼ均一。日本や中国の原油はそれぞれ違う。
iso-octane で沈殿させて、泥のようなものがたまったところから DNA 抽出キットで DNA を拾う (菌そのものを拾って培養するわけじゃなくて、配列だけ拾う)。
16S rRNA から菌種を推定。アルカンを分解する菌、芳香族を分解する菌などが出てくるが、シアノバクテリアや耐熱性の菌などが出てくる上に、産地でそれぞれまったく異なった population になる。
世界中の原油から出てくる菌は石油の精製に関わっている?
中国の砂漠とか台湾の山とか、ヨルダンの温泉からとったシアノバクテリアに類似する rRNA が出てきたりしてる。
なんかすげーなー。

原油を微生物が作った、と証明している人はいないから、まず菌をとって、石油ができるところを証明しないといけないのでは? → おっしゃる通りですが、それは無理だと思うわけで、いまのやりかたでできることもあると思う。
真核生物といっしょにいる可能性の高い共生 (あるいは病原) 微生物に偏っているとか、そういうことはある? → まだ何もわかっていません。
PCR プライマー的に古細菌などはみている? → 1種類しか見つかっていませんが、見てはいます。
炭化水素の代謝とかは? → メタゲノムなのでぜひやりたいが、なにぶん収量が少ないので。
[ 9: 偽遺伝子が明らかにしたシロアリ腸内共生未培養新門細菌の適応進化 ]
シロアリの腸には6万の原生生物と1千万の腸内細菌!
これらが木材のセルロースを分解したり、窒素固定をして窒素を補ったりする (いくら木を食べても、タンパク質不足なので)。
でも、ほとんどが培養されていないので何をしているかわからない。
Trichonympha agilis という原生生物が重要。で、こいつは中に細菌をたくさん飼っている (新門である Termite Group 1: TG1)。Trichonympha も TG1 も培養できないが、たくさんとってきて TG1 細菌 Rs-D17 の環状ゲノムを決定した。こいつは 121 個の偽遺伝子を持っている! 偽遺伝子があるので、細胞内共生の様子を探ることができる。ゲノム長は 1.1Mbp
偽遺伝子はおもに DNA 修復、細胞壁合成、トランスポート、defense など。環境応答系はそもそも遺伝子が欠落している。
dnaA (染色体複製開始因子) が pseudogene になっている!
同じように dnaA が欠損しているのが、昆虫の細胞で共生する奴らなので、どうやらそういう関係にあると dnaA が欠落するらしい。recA などの recombination 系を使ってなんとか複製している、といわれている。
ペプチドグリカン合成系 (細胞壁つくる) はほぼ残っているように見えたが、ほとんどが偽遺伝化している。偽遺伝子で残っているということは、共生し始めてそれほど時間がたっていない?
制限酵素遺伝子群は26セットもあるけど、2セットを除いてほとんど偽遺伝子になっているか、認識ユニットだけで制限酵素ユニットが落ちている。
CRISPR システムを持っているから、ファージの攻撃が多い環境だった?
アミノ酸合成や補因子合成の遺伝子は生きているので、シロアリにアミノ酸や補因子を供給する役割を果たしている?
偽遺伝子になるか欠落するかはどう違うの? → よくわからないんだけど、いらなくなって deletion しようと思っても、修復系の遺伝子がまだ生きているとけっこう残っちゃうのかな、と。
高等シロアリには、共生原生生物はいない (細菌はいるけど)
[ 10: シロアリ共生原生生物のメタトランスクリプトーム解析 ]
嫌気性の奴らが多く、培養は大変。材料はオオシロアリ。
原生生物は Parabasalia と Oxymonadida が主。これらをまとめて EST 解析。
total RNA から rRNA を抽出して構成生物種を解析。
mRNA だけを抽出して、完全長 cDNA library を作成。平均 573bp, 合計 140Mbp.
micro manipulator を使って特定の原生生物の細胞をとり、cDNA library をつくることもやってみた。
actin, alpha/beta-tublin など、代表的な遺伝子をしらべてみると、かなり diversity が大きい。
木質分解酵素の種類やタイプも、原生生物ごとに異なる。
結晶性・非結晶性セルロースの分解系がかなり発現している。こいつらはカビ・キノコのセルラーゼと違って、セルロースへの binding site をもっていない。
メタゲノム的には何かやってない? → どうしても細菌と (ゲノムサイズの大きな) 原生生物が混じっちゃうので、まだやってません。
[ 11: 持続可能型社会への貢献遺伝子データベース ”膨大な環境由来メタゲノム配列からの有用遺伝子探索” ]
メタゲノム配列からの有用遺伝子探索。
長浜バイオ大の人のスライドっていつもこんな感じな気がする。
自分の学生がやったら怒るけど、でも、これはこれでけっこう好き。


メタゲノムでみつけた遺伝子の大半は機能注釈がついていない。これを、
– 学生にテーマをみつけさせて
– Swiss-Prot とかで関係する遺伝子をみつけさせ
– それに近いメタゲノムな遺伝子を blast で釣ってくる (もちろん、ほかにも curation のプロセスを踏んでいる)
といった感じでデータベースに登録していく。
[ 12: メタゲノムからの未知有用遺伝子スクリーニング法の開発 ]
– 活性ベース: クローンを作って、酵素活性でスクリーニング
– 配列ベース: 酵素の配列をもとに選抜し、実際に発現させる
– SIGEX (Substrate Induced Gene Expression): 酵素活性をはかるのではなく、プロモータが on になるかを (GFP とかで) 見て釣ってくる。かっこいい。でも、ウソなのを引いて来ちゃうこともあるので注意。
[ 13: プロファージ遺伝子を含む原核生物ゲノムからの遺伝子予測 ]
メタゲノム遺伝子予測に向けた metagene というソフトウェア (高木先生のところで作った)
– GC% から2連コドン頻度を推定して、予測に利用 (生物種が不明な場合の遺伝子予測に有効)
– Bacteria-Archia の推測を、2連コドン頻度をもとに行う
など。入力の生物種をきにせずに遺伝子を予測できる。完全長でも、700bp にちぎった断片配列でもイケる。200bp とかになると、さすがにしんどい。あんまり短く読んでくるシーケンサだと・・・
で、Bacteria-Archia に加えて Phage も入れた。最新版は MetageneV.
O157 のようなファージ由来の外来性遺伝子に対する感度が向上。
極端ならいいけど、GC% が 50% くらいでどっちつかずだと怖くない? → テストデータを見る限りではうまくいっている。
ゲノム微生物学会 Mar.06 / oral session 1
[ 1: アオコ形成シアノバクテリアMicrocystis aeruginosaのゲノム構造解析 ]
アオコはいろいろな微生物が作る。
単細胞性の奴が多い。Microcystis aeruginosa とか。
毒素 (ペプチドとか) を作る。非運動性。水に浮いている状態で群体をつくる。
群体はバイオフィルムというか、多糖のかたまりのようなもの。
ゲノムは 5.8Mbp, 6312 protein coding gene, 2 rRNA set, tRNA 42
転移性遺伝因子 (IS, MITE) が多い。
Synechocystis に似ているが、似ていない COG category もあるぞ。
Synechocystis は群体をつくらない。多糖合成が違い?
いまのところ Mycrocystis のゲノムからは、多糖合成のところはよくわかってない。
似ていないの:
– IS と MITE がたくさんある (repeat sequence が多い、組み替えが頻繁に起きる)
– Signal transduction mechanism が半分くらいに減っている。わりと簡単
– Cell motility: まあ、運動しないのでね
– 二次代謝産物の transport, catabolize 関係
– Replication recombination and repair: 制限酵素とかたくさん持っている!
[ 2: NITE・バイオ有用シアノバクテリアSpirulina (Arthrospira) platensis NIES-39のゲノム解析 ]
NITE の藤澤先生。
古代メキシコの時代から食品だとか添加物として。タンパク質含有量が 65%.
健康補助にもつかわれている。
アフリカのチャド湖で採取され、国内で維持されてきた株をゲノム解析。
Contig が 19 本の状態。contig 総延長が 6.69Mbp, 推定ゲノムサイズは 6.78Mbp. GC contents 44.3%.
2 rRNA set, 42 tRNAs. Tandem repeat (74mer とか) が多いのでなかなか contig のギャップが埋まらない。
Group II intron のためにゲノム内の相同領域が多い (dotplot だと真っ赤な状態) のも原因。
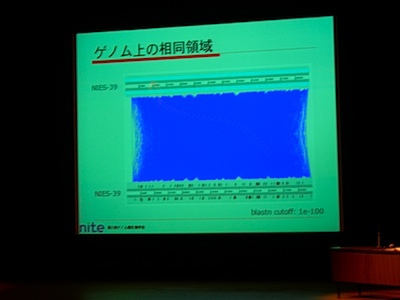
71 ORF coding group II introns + 79 non ORF coding group II introns. 前者から ORF を取り除くと、両者は非常によく似た配列。
88 Rfam:RF00029 (RNA doman V, VI) RNA motifs.
それだけ内部ホモロジが高いとゲノムが不安定にならない? → どうやって安定化しているかは謎です。
[ 3: グラム陽性嫌気性球菌Finegoldia magna ATCC 29328株の全ゲノム解析 ]
Peptostreptococcus 属の一菌種。皮膚や粘膜の常在菌。
アルブミン結合タンパク (抗貪食性に寄与) やコラーゲン接着因子、セリンプロテアーゼなど、病原性が高い感じのものをたくさんもっているが、ゲノムはあまり解析されていない。
1.79Mbp, 32.3% GC content, 1631 ORFs…
IS はひとつしかないので、外来遺伝子とかはあんまりなくて安定していそう。
糖をほとんど分化できないが、アミノ酸代謝は合成も分解もあり。TCA サイクルは acetate でとまっており、嫌気性代謝経路が著しく欠落。
菌体の表面にアルブミン結合タンパクを持っており、食われないようになっている。
4つのアルブミン結合タンパクを発見。4つとも GA module (アルブミン結合部位) を持っている。
染色体の sortase の数は他の菌とさほどかわらないが、プラスミドにはたくさん (でも、この株特有の現象かも?)。
(sortase や基質の同定は in silico)
sortase ってなに? (アホですいません。電気屋なのでね…)
アルブミン結合タンパクが抗貪食性に寄与するメカニズムは? → 分子レベルではまだわかってません
[ 4: α溶血性レンサ球菌Lactococcus garvieaeの比較ゲノム解析によるブリ属魚類への病原性遺伝子の探索 ]
ブリの病気。敗血症と膿瘍と両方。
Lactococcus なので、チーズスターターと近い (でも、ヨーグルトスターターの近縁にも危ない奴がいるぞ!)
ウシ敗血症でみつかった菌。でも、健常動物の腸管や生野菜なんかからも取れる。
ブリに対して病原性のある株 (Lg2 株) とない株 (ATCC49156株) が存在。莢膜があるものが毒性あり。
このふたつは非常にきれいに synteny がでるが、真ん中がちょこっと逆胃している。
Lg2株も、TTC 添加 EF 寒天培地で増やしてやると弱毒化。莢膜合成遺伝子群にフレームシフトが起きている。
野菜由来の株はブリに対する毒性なし。莢膜生成遺伝子群がない。
莢膜と hemolysin の存在が類似している。hemolysin っぽい遺伝子はふたつあり、毒性がある株にもない株にもまったく同じ配列で存在している。
莢膜は病原因子だけど毒素ではなさそう。生体侵入時の菌体の防御系?
莢膜はなにでできてる?→多糖だと思うんだけど、まだよくわからない。
[ 5: Streptococcus mutansゲノム解析に基づく種レベルでの進化機構の解析 ]
むしばです。
でも、もっと危ない病気も起こすらしい。
日本で分離された S. mutans NN2025 のゲノム解析。2Mbp くらい。
ファージの残渣がほとんどないので、ファージ抵抗性がある模様。

全体的にぐるっと逆位する rearrangement が多いっぽい。
解析に Mauve 使ってる!!! がびーん。Mauve でおもしろそうなブロックをしぼって long PCR したり。
Mauve で出てくる gap は IS や Tn で、外来性遺伝子はこのあたりで獲得していると思われる。
CRISPR-1, CRISPR-2(Clustered regulated interspaced short palindromic repeats?) が外来遺伝子に対する抵抗性を持つ。

[ 6: 腸管出血性大腸菌(O26, O111, O103)の全ゲノム解析 ]

EHEC: 腸管出血性大腸菌 (O157, O26, O111, O103 など)
O157-specific genes: 1632 (ゲノム中に散在、外来性と思われる)
3型分泌装置、志賀毒素 (Stx1 & Stx2) などをもつのが、EHEC のくくりで、系統樹的な違いではない。
Prophages: 18 (lambda-like が多い。Stx1,2 も)
Integrative elements: 6
O157 vs K12 でたくさんドットが出るところは、prophage だったりするところが多い。
EHECのなかでは O157 だけが系統樹的にだいぶ遠い。別々に毒性を獲得しながら平行進化してきたと考えられてきた。
配列を調べてみると O157 とほかの EHEC はかなり近いんだけどね…
(すみません、途中で意識が飛びました)
[ 7: 結核菌 (Mycobacterium tuberculosis) 北京型ファミリ内における微小分子進化 ]
繰り返し配列の変化。
ゲノムの安定性が高く、水平伝搬とかが少ないので系統樹が書きやすい。
北京型結核菌は東アジア (China, Korea, Japan) あたりの結核菌のほとんどを占める。

結核菌には VNTR (縦列反復配列多型) が多数 (十数個) あり、型別分類に利用される。
VNTR 領域を PCR して電気泳動して分類できる。

MST (minimum spanning tree) で、VNTR 型別から系統樹を作成。300株以上で、既存の SNPs による分類とも重なる。
VNTR 変異は、系統分類の分子マーカーとして使える。
Acrobat 7 Standard vs. Leopard
時間がなくて、MacBook で Keynote から Acrobat にデータを流して PDF にしようとしたら、Distiller が死んじゃって、うまくいかなかった。Leopard ではうまく動かないみたい。8.0 買うかなー。
しんどいねぇ。
mixi
4/1 から mixi の利用規約が改正される模様。
第21条に「本規約は法令よりも優先」みたいな記述があるんだけど、これはどういうことだろう… ここは日本ですよ。国内の企業が国内の人間に対して提供するサービスは、当然国内の法律が適用されるはずですが。
しかも、23条には「準拠法は日本の」とか書いてあるし。法令より優先するなら準拠法なんていらないでしょ。
でもって、附則には「施行前にユーザによって行われた行為も対象」みたいなことが書いてあるわけですが、なんか、「法令よりも優先」とか書かれているものに、こういう附則がついているとものすごく不気味。21条のそれがなければ、別にかまいませんけれど。
これって、ちゃんと法律のことわかってるひとが書いたのかしら?
法治国家をナメてるのか、それとも僕ら素人にはよくわからない難しい法律のお話があるのかしら。無効確認訴訟とか起きそうだな。
mixi ってすてきな会社だな、と思ってたのだけど…
何かを壊された気分。
みのまわり
仕事ばっかりだが、たまには身の回りのこともせねばならない。
というわけで今日は、
– 昼間で寝た。じゃないと身体がもたない
– ひたすら実装した (身の回りのことではないな)
– 愛用の、スターバックスのタンブラーを歯ブラシと漂白剤で洗った (普段から気をつけてても、どうしても茶渋とかたまるからねー)
– もろもろの請求を ANA カードに一本化
した。いちばん進んだのは実装かもね。それって仕事じゃん。むふ。