[ 1: アオコ形成シアノバクテリアMicrocystis aeruginosaのゲノム構造解析 ]
アオコはいろいろな微生物が作る。
単細胞性の奴が多い。Microcystis aeruginosa とか。
毒素 (ペプチドとか) を作る。非運動性。水に浮いている状態で群体をつくる。
群体はバイオフィルムというか、多糖のかたまりのようなもの。
ゲノムは 5.8Mbp, 6312 protein coding gene, 2 rRNA set, tRNA 42
転移性遺伝因子 (IS, MITE) が多い。
Synechocystis に似ているが、似ていない COG category もあるぞ。
Synechocystis は群体をつくらない。多糖合成が違い?
いまのところ Mycrocystis のゲノムからは、多糖合成のところはよくわかってない。
似ていないの:
– IS と MITE がたくさんある (repeat sequence が多い、組み替えが頻繁に起きる)
– Signal transduction mechanism が半分くらいに減っている。わりと簡単
– Cell motility: まあ、運動しないのでね
– 二次代謝産物の transport, catabolize 関係
– Replication recombination and repair: 制限酵素とかたくさん持っている!
[ 2: NITE・バイオ有用シアノバクテリアSpirulina (Arthrospira) platensis NIES-39のゲノム解析 ]
NITE の藤澤先生。
古代メキシコの時代から食品だとか添加物として。タンパク質含有量が 65%.
健康補助にもつかわれている。
アフリカのチャド湖で採取され、国内で維持されてきた株をゲノム解析。
Contig が 19 本の状態。contig 総延長が 6.69Mbp, 推定ゲノムサイズは 6.78Mbp. GC contents 44.3%.
2 rRNA set, 42 tRNAs. Tandem repeat (74mer とか) が多いのでなかなか contig のギャップが埋まらない。
Group II intron のためにゲノム内の相同領域が多い (dotplot だと真っ赤な状態) のも原因。
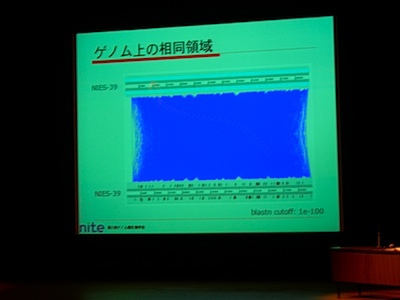
71 ORF coding group II introns + 79 non ORF coding group II introns. 前者から ORF を取り除くと、両者は非常によく似た配列。
88 Rfam:RF00029 (RNA doman V, VI) RNA motifs.
それだけ内部ホモロジが高いとゲノムが不安定にならない? → どうやって安定化しているかは謎です。
[ 3: グラム陽性嫌気性球菌Finegoldia magna ATCC 29328株の全ゲノム解析 ]
Peptostreptococcus 属の一菌種。皮膚や粘膜の常在菌。
アルブミン結合タンパク (抗貪食性に寄与) やコラーゲン接着因子、セリンプロテアーゼなど、病原性が高い感じのものをたくさんもっているが、ゲノムはあまり解析されていない。
1.79Mbp, 32.3% GC content, 1631 ORFs…
IS はひとつしかないので、外来遺伝子とかはあんまりなくて安定していそう。
糖をほとんど分化できないが、アミノ酸代謝は合成も分解もあり。TCA サイクルは acetate でとまっており、嫌気性代謝経路が著しく欠落。
菌体の表面にアルブミン結合タンパクを持っており、食われないようになっている。
4つのアルブミン結合タンパクを発見。4つとも GA module (アルブミン結合部位) を持っている。
染色体の sortase の数は他の菌とさほどかわらないが、プラスミドにはたくさん (でも、この株特有の現象かも?)。
(sortase や基質の同定は in silico)
sortase ってなに? (アホですいません。電気屋なのでね…)
アルブミン結合タンパクが抗貪食性に寄与するメカニズムは? → 分子レベルではまだわかってません
[ 4: α溶血性レンサ球菌Lactococcus garvieaeの比較ゲノム解析によるブリ属魚類への病原性遺伝子の探索 ]
ブリの病気。敗血症と膿瘍と両方。
Lactococcus なので、チーズスターターと近い (でも、ヨーグルトスターターの近縁にも危ない奴がいるぞ!)
ウシ敗血症でみつかった菌。でも、健常動物の腸管や生野菜なんかからも取れる。
ブリに対して病原性のある株 (Lg2 株) とない株 (ATCC49156株) が存在。莢膜があるものが毒性あり。
このふたつは非常にきれいに synteny がでるが、真ん中がちょこっと逆胃している。
Lg2株も、TTC 添加 EF 寒天培地で増やしてやると弱毒化。莢膜合成遺伝子群にフレームシフトが起きている。
野菜由来の株はブリに対する毒性なし。莢膜生成遺伝子群がない。
莢膜と hemolysin の存在が類似している。hemolysin っぽい遺伝子はふたつあり、毒性がある株にもない株にもまったく同じ配列で存在している。
莢膜は病原因子だけど毒素ではなさそう。生体侵入時の菌体の防御系?
莢膜はなにでできてる?→多糖だと思うんだけど、まだよくわからない。
[ 5: Streptococcus mutansゲノム解析に基づく種レベルでの進化機構の解析 ]
むしばです。
でも、もっと危ない病気も起こすらしい。
日本で分離された S. mutans NN2025 のゲノム解析。2Mbp くらい。
ファージの残渣がほとんどないので、ファージ抵抗性がある模様。

全体的にぐるっと逆位する rearrangement が多いっぽい。
解析に Mauve 使ってる!!! がびーん。Mauve でおもしろそうなブロックをしぼって long PCR したり。
Mauve で出てくる gap は IS や Tn で、外来性遺伝子はこのあたりで獲得していると思われる。
CRISPR-1, CRISPR-2(Clustered regulated interspaced short palindromic repeats?) が外来遺伝子に対する抵抗性を持つ。

[ 6: 腸管出血性大腸菌(O26, O111, O103)の全ゲノム解析 ]

EHEC: 腸管出血性大腸菌 (O157, O26, O111, O103 など)
O157-specific genes: 1632 (ゲノム中に散在、外来性と思われる)
3型分泌装置、志賀毒素 (Stx1 & Stx2) などをもつのが、EHEC のくくりで、系統樹的な違いではない。
Prophages: 18 (lambda-like が多い。Stx1,2 も)
Integrative elements: 6
O157 vs K12 でたくさんドットが出るところは、prophage だったりするところが多い。
EHECのなかでは O157 だけが系統樹的にだいぶ遠い。別々に毒性を獲得しながら平行進化してきたと考えられてきた。
配列を調べてみると O157 とほかの EHEC はかなり近いんだけどね…
(すみません、途中で意識が飛びました)
[ 7: 結核菌 (Mycobacterium tuberculosis) 北京型ファミリ内における微小分子進化 ]
繰り返し配列の変化。
ゲノムの安定性が高く、水平伝搬とかが少ないので系統樹が書きやすい。
北京型結核菌は東アジア (China, Korea, Japan) あたりの結核菌のほとんどを占める。

結核菌には VNTR (縦列反復配列多型) が多数 (十数個) あり、型別分類に利用される。
VNTR 領域を PCR して電気泳動して分類できる。

MST (minimum spanning tree) で、VNTR 型別から系統樹を作成。300株以上で、既存の SNPs による分類とも重なる。
VNTR 変異は、系統分類の分子マーカーとして使える。