応用ゲノム。
[ 38: 塩類集積環境の重金属浄化に有用なセルフクローニング型アーミング Halomonas elongataの作製 ]
塩性化・アルカリ化・重金属の濃縮などが同時に起こる場合の浄化。
H. elongata は金属結合ドメインをもっており、それを外膜リポタンパク質にくっつけることで、細胞表層に呈示するようにする。
外来遺伝子を導入せずに、相同組み替えだけで実現。
1. 高塩濃度条件 (3% NaCl, 18% NaCl) で安定して機能する外膜リポタンパクの選定。
2. 高発現させたときに細胞が凝集しないものをえらぶ
3. そのリポタンパクが欠失しても (これから機能を変えちゃうので) 塩ストレスで死滅したりしないかを確認
Cu 選択的な浄化能を示すものができた。
ドメインを多重化することで、さらにパワーアップ!
[ 39: 枯草菌ゲノム縮小株のトランスクリプトーム解析 ]
枯草菌の高いタンパク質分泌能力と、容易な操作性は工業用途にも便利。
だけど、ストレス反応みたいな、いらない機能もあるわけで、それらを削除することで、物質生産に特化したものをつくりたい。
先行研究で、7.7kbp の外来性遺伝子領域を削除したり、必須遺伝子を同定した研究はあるが、生産性向上に寄与したかどうかの評価はない。
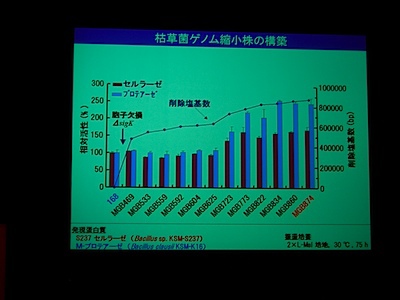
874kbp を削除し、セルラーゼ・プロテアーゼの活性を 200% 向上させることに成功。増殖は若干遅いが、定常期においても高い生産性を維持している。
胞子形成に至るシグマ因子の発現は遅れている?
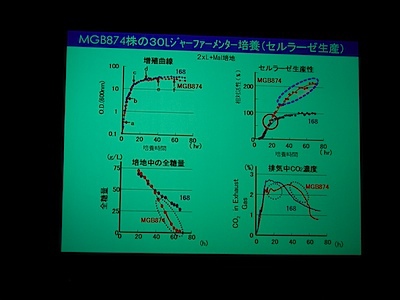

削減株が培養後期でも活発ということは、後期での viability が低下することにならない? → CO2 排出量が減っていないんで、よさそう。
成長をとめて増殖の準備をしようとするところがなくなる、と思うのですが → 後期の細胞の維持もちゃんとやっていそう。溶菌に関するところは削除されている。
セルラーゼ・プロテアーゼ以外の生産性向上については? → 分泌が律速になってあがらないもの (アミラーゼなど) があることもあるが、そこをいじってやれば何とかなると思う。
[ 40: DNAマイクロアレイデータ解析に基づいたエタノールストレス耐性を示す酵母菌株の創製 ]
実験室酵母と、ストレス耐性を有する醸造酵母(協会7号: IFO2347) の違いは?
ストレス耐性遺伝子の発現はエタノール添加前後で違う?
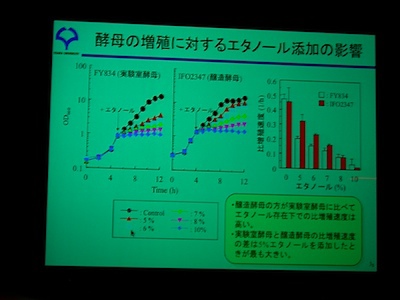
ストレス耐性遺伝子は、なにもない状態でも醸造酵母のほうがたくさん発現している。
実験室酵母でも、tryptophan の取り込みに関わる遺伝子と、培地への tryptophan 添加でエタノール耐性が出る。
実験室酵母が1倍体で協会7号が2倍体なのはどうかと・・・→ すみません、いろいろありまして。
そうか、酵母菌って2倍体もいるのよね。たいへんだ。
[ 41: 乳酸耐性酵母の分子育種工学 -欠失により乳酸感受性を付与する出芽酵母遺伝子の網羅的同定と機能解析- ]
ポリ乳酸がプラスチックとして注目されており、その原料の乳酸を生産させるのに出芽酵母が注目されている。乳酸耐性をうまくもたせることができれば、中和処理なしで大量生産ができるようになる!
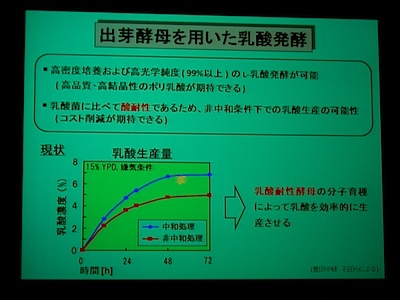
乳酸耐性には液胞や細胞内輸送に関係する遺伝子が重要。
– 破壊でも乳酸耐性を引き起こすことができる
– 多コピーで乳酸耐性を得ることもできる。ストレス応答などの遺伝子をプラスミドにいれて overexpression することで乳酸耐性化。安定して動かすために、染色体上で高発現プロモーターとくっつけて成功。
[ 42: カンジダ酵母における病原性ゲノム機能学(網羅的遺伝子発現制御株の構築と応用) ]
S. Ce の必須遺伝子群との ortholog をもとに、必須遺伝子群を同定。800-1000遺伝子くらい?
Tet 株を使っている。マウスに感染させたあとで、飲み水で発現を制御できるので便利。
これをもとに高真菌薬をつくりたい。必須遺伝子群のなかで、ヒトには存在しないものを標的にすることで薬効が高く、副作用の少ないものを狙う。病原真菌に高く保存されている遺伝子に絞り込むことで広い範囲に効くようにもしたい。
5,300 genes / 150 screened in silico / 10 genes in vitro 。実験頑張ってます。ほんとは10個じゃなくて、もうすこしやりたいですが・・・
抗真菌ペプチド医薬。ペプチドを環状化することで安定化させたい。インシリコで分子進化させたものをいくつか実際につくって、テストしている。
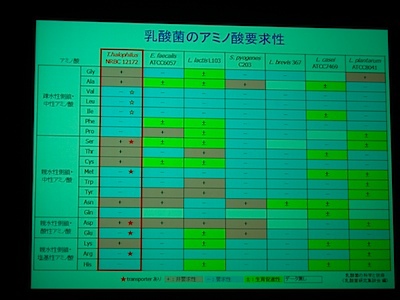
[ 43: 醤油乳酸菌Tetragenococcus halophilus NBRC 12172株のゲノム解析 ]
IS が多くて、他の乳酸菌と比べるとゲノム構造はあまり保存されていない。
でも、COG 分類を眺めると、遺伝子は大差ないことがわかる。
TCA サイクルは一部が欠けている、アミノ酸合成系の一部が欠けている、といった特徴。これはほかの乳酸菌とかわらない。
大事なのは塩耐性。
Choline → Glycine betaine の合成系や Na / K の排出 / 取り込み機構 が重要な重要っぽい。
Na 輸送に際して ATP を作れる! これも塩耐性に貢献しているはず。
ほかの耐塩性乳酸菌とはくらべた? → ほかのゲノムが公開されたばかりなので、まだ比べていない。