[ DNA はどのように折りたたまれているのか? ]
森下先生@東大。
源氏物語: 原本は存在しなくて、写本がたくさんあり、それぞれ巻数が違ったり indel とかがある → ゲノムの染色体の本数とか配列の違いに似ている?
ヒトのゲノムは 10 時間で複製される。50塩基/秒で、100並列。
祖先ゲノムがどうだったか、ということを知るのに、ヒト・チンパンジーとホヤ・ナメクジウオだけではなくて真ん中の魚類くらいのが必要だった。
脊椎動物の祖先ゲノムは10本くらいの染色体があったのではないかと推定しており、ヒトに至るには2度のゲノム重複が起きている。魚類はさらにもう一度。
染色体が物語の一巻だとしたら、ページに対応するのがヌクレオソーム構造。
プロモーター結合がページを開くことに、メチル化がページを閉じることに例えられる。
(染色体をコピーするときはヒストンに巻きついた状態で複製され、体細胞の分裂の際にはメチル化の情報も伝わっているっぽい、のだそうです。)
メダカ+Solexa で調べてみた結果、ヌクレオソーム構造を取っているところとそうでないところで、塩基置換率が違う (ヌクレオソーム構造を取っているところでは、ヌクレオソーム構造を保存する方向に塩基置換が起きている。10年くらい前の酵母の研究では、ヌクレオソーム構造の中心ではリンカーの部分に比べて修復率が落ちているので、そっちが問題なのかも) ことがわかった。おもしろいな。
コンピュータはだいたい 2x in 18month なのが、sequencer は 2x in 8month なのでやばいね、というお話も。そうなんです…
Solexa は 1bp/h くらいで超並列だけど、Pacific bio のは 4bp/s くらい。ただ、後者は並列化が課題みたい。
中国はすごい勢いで Solexa GA とかを購入して頑張っているそうだ (台数としては、日本全体とあんまりかわらないけど、日本は分散しているのが問題かな。)。うおー。がんばらないとなあ。
[ Wiki によるデータベースと研究成果の発信 ]
有田先生@東大。
計測と解析は速くなった。でもその真ん中にある整理 (curation) は?
科学は知識の積み上げだから、成果が再利用されないと意味がない。でも、論文も学会発表も報道発表も、再利用には向いていない。
我々はデータ中心という新しいパラダイムに直面している。一般人は科学を信じ、期待しているのに、科学者は一般人に理解してもらうことを放棄。科学者がデータ中心の概念に対応できていないのは問題で、いつまで雑誌崇拝がつづくのか?
subscription fee が上がっている背景には、投資ファンドが雑誌の出資元を持っていることも影響している。
有田先生 Wiki はすでにいくつかの学会の公式なデータベースとして認定していただいているそうで、成果公開というのは大変重要なプロセスにおけるこういう活動は非常に重要だと思う。
数式とか理論的なものはいいけど、ゲノムとかそういう大規模なデータはどうやったらいいと思いますか (森下先生) → Wiki は backend が RDB なので、なにかそういうものを考え直さないといけない。いずれにしても Web ベースでやれる仕掛けは必要。
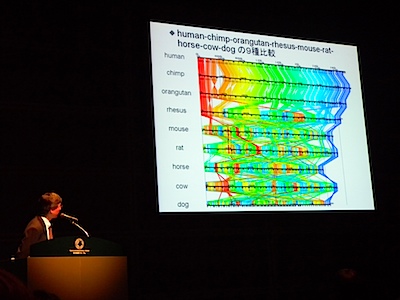
[ 多生物種のゲノムを高速に比較する並列システムの開発 ]
榊原先生@慶應大。
質疑だけメモ。
– マウス・ラットでは再編成が起こりやすい系統があるようですがそういうのと比べてみると面白いかもしれません。
– X染色体はヒトとチンパンジーでかなり保存されているがY染色体はシャッフルされている、という話がでているけれど、どうでしょう。 → Y のほうはまだアセンブリがきれいに進んでいない生物が多いので比較ができないのが現状です。
– NIG とか UCSC で visualize できなくて Murasaki & GMV でできるものは? 入力フォーマットは一般的なもの? → お答えしておきました。まさか回答させられるとは・・・